
génomique ENVIRONNEMENTALE (GE)
Contacts :
Responsables :
Tristan Lefébure (tristan.lefebure@univ-lyon1.fr)
Annabelle Haudry (annabelle.haudry@univ-lyon1.fr)
Mylène Hugoni (mylene.hugoni@univ-lyon1.fr)
Objectifs de la formation
Nous savons maintenant que notre compréhension de la biodiversité reste très parcellaire à l’échelle globale. En effet, des environnements sont difficilement accessibles (eg. fonds océaniques),
des branches entières du vivant restent inexplorées (eg. archées), la plupart des associations symbiotiques sont peu connues (eg. microbiomes), et même pour les pans de biodiversité les mieux
connus, nos outils de suivi de la biodiversité sont mal adaptés face aux défis des changements actuels et à venir. Dans ce cadre, l'essor récent de la génomique, et en particulier les
possibilités de séquencage haut-débit, offre une large palette d’outils et de méthodes pour décrire la biodiversité, étudier son fonctionnement et comprendre son évolution. L’objectif du parcours
GE est de former des étudiant.e.s aux outils de la génomique pour répondre aux défis écologiques actuels.
Le contenu pédagogique du parcours GE@lyon vise à apporter et combiner les compétences scientifiques de trois disciplines différentes : l’écologie, la génomique et la
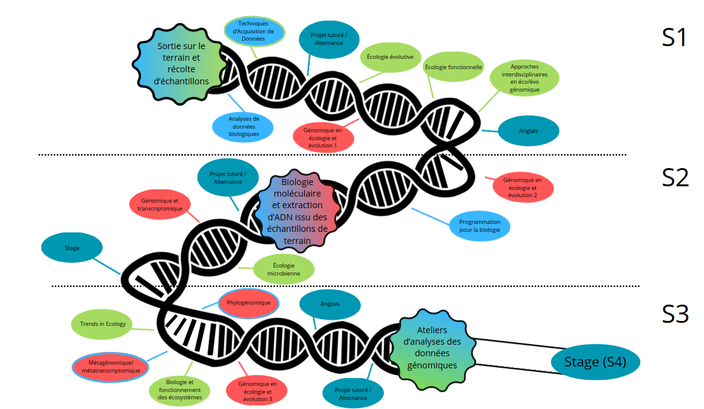
bioinformatique. Ces disciplines seront toutes trois abordées dans des enseignements du S1, S2 et S3; dans un effort de continuité pédagogique. Un objectif fort du parcours est la prise
d’autonomie pour conduire des projets de génomique environnementale. Cette autonomie sera acquise lors de stages et de projets tutorés d’une part, et d’autre part au travers de trois UE qui se
succéderont (terrain et échantillonnage au S1 ; préparation des échantillons, biologie moléculaire et séquençage au S2 ; analyses bioinformatiques et
communications des résultats sous forme d’article au S3) apportant ainsi une expérience complète aux étudiants en menant à bien de manière collaborative un projet de recherche de son
initiation à sa valorisation. Dans toutes les UEs, l’accent sera mis sur la compréhension et la mise en pratique des approches enseignées (TD, TP). Les compétences bio-informatiques nécessaires
pour mener des projets de génomique environnementale seront apportées aux étudiant.e.s au cours du master (pas de pré-requis en informatique pour rentrer dans ce parcours).
débouchés
Le parcours GE a pour objectif de former des chercheurs, des chercheuses et des expert.es capables de construire et de mener à bien un projet de génomique environnementale dans son entièreté : la formulation d’une problématique d’écologie, la mise au point d’un protocole d’échantillonnage, le choix des outils moléculaires, statistiques, et bioinformatiques et l’analyse, l’interprétation et la communication des résultats au regard de la problématique. Les possibilités d’emplois en sortie de master sont diversifiées car ces compétences intéressent différentes structures :
-
Doctorat en écologie, évolution, génomique…(p.ex. au sein de l’école doctorale E2M2)
-
Ingénieur.e sur des projets de recherche
-
Ingénieur.e sur des plateformes de génomique environnementale (publique ou privée)
-
Bureaux d'études en génomique environnementale ou environnement
-
Entreprise du biomédicale...
L’atout majeur du parcours Génomique Environnementale (GE) consiste en une approche globale, avec l’apprentissage des compétences permettant une approche par projet de résolution de questions
d’écologie intégrant des outils de génomique.
Les compétences bio-informatiques acquises au cours de ce parcours seront fortement valorisables à l’issue du master, quel que soit le débouché choisi.
contenu de la formation
Semestre 2
Le semestre 2 renforce les compétences en génomique, en bioinformatique, en écologie microbienne, en biologie moléculaire (préparation et séquençage des échantillons collectés au S1); et enfin
met les étudiant.es en situation d’autonomie sur un projet tutoré et un stage.
Approfondissement des connaissances en Génomique pour l’Ecologie et l’Evolution : dynamique d'évolution des génomes (impact de transferts horizontaux, effets de la recombinaison,
insertion d’éléments mobiles) et reconstruction de l’histoire évolutive de populations.
Une UE de Biologie Moléculaire afin de développer des approches allant de la génomique à la métagénomique: préparation et séquençage des échantillons prélevés au S1.
Un enseignement spécialisé sur la Microbiologie Environnementale.
Une UE de Programmation pour la Biologie (apprentissage de python, accès à un serveur distant, manipulation de gros jeux de données), complétée par une UE de bioinformatique pour
apprendre à traiter des données de Génomique et Transcriptomique.
Mise en autonomie sur un projet tutoré ainsi qu’un stage de 7 semaines.
master 2
Approfondissement des connaissances en Génomique en Ecologie et Evolution.
Analyse bioinformatique des données de séquencage obtenues au S2 et communication des résultats dans l’UE Ateliers d’analyses en génomique
environnementale.
Ouverture scientifique sur des grandes questions en écologie – séminaires de recherche, en anglais, par des spécialistes français et étrangers.
Présentation de la métagénomique / métatranscriptomique comme discipline émergente: portée, limites et challenges; et analyse de données.
Choix d’une UE parmi 2 options : Phylogénomique (reconstruction d’arbres, écologie évolutive) ou Biodiversité et Fonctionnement des Écosystèmes (écologie
trophique et fonctionnelle).
Le S4 sera consacré entièrement au stage de longue durée (5 mois), effectué en laboratoire de recherche ou en entreprise.

